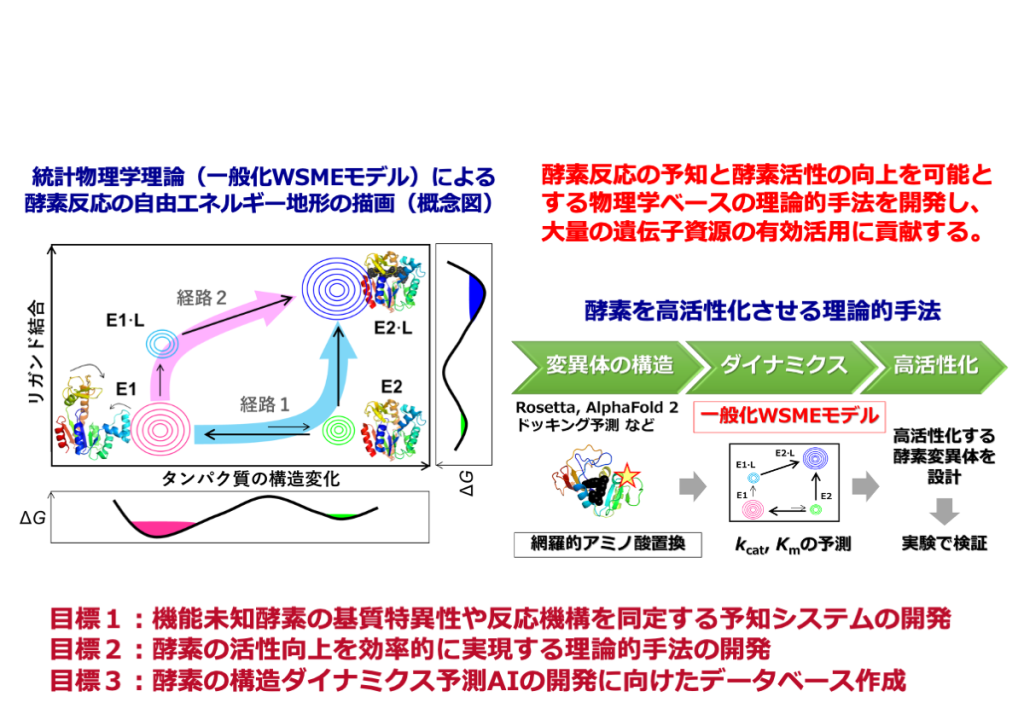
酵素反応の予知と酵素活性の向上を可能とする物理学ベースの理論的手法の開発
研究者紹介

研究代表者
新井宗仁
東京⼤学大学院
総合文化研究科
研究概要
大量の遺伝子資源を有効活用するためには、【課題①】「機能未知酵素の基質特異性や反応機構を同定する予知システムの開発」と、【課題②】「酵素の活性向上を効率的に実現する理論的手法の開発」が必要である。しかし酵素反応は複雑な構造変化を伴うため、これらの理論予測は極めてチャレンジングである。我々は最近、これらを実現する上でのブレイクスルーとなりうる物理学理論(一般化WSMEモデル)の開発に成功した。この理論は、小型タンパク質のフォールディング反応機構を説明できる統計力学理論(WSMEモデル)を、あらゆるタンパク質にも適用できるように拡張した理論であり、タンパク質の立体構造さえ得られれば、その自由エネルギー地形(構造ダイナミクス)や、酵素反応のkcat,Kmを予測でき、酵素反応機構の理論的・包括的描画が可能になる。
そこで、この物理学ベースの理論を応用して、上記課題①②と【課題③】「酵素の構造ダイナミクス予測AIの開発に向けたデータベース作成」を目指す。課題①については、機能未知酵素の予測構造から、リガンド結合強度やkcat, Km、および反応機構までを包括的に予測し、機能未知酵素の機能やメカニズムを推定できる理論的手法を開発する。課題②については、酵素変異体の自由エネルギー地形からkcat, Kmを予測し、高活性化変異体を効率的に設計可能な画期的手法を開発する。また、いくつかのモデル酵素に本手法を適用し、実験によって妥当性を検証する。課題③については、酵素の構造ダイナミクスを深層学習によって予測可能とするために、一般化WSMEモデルで様々な酵素の構造ダイナミクス(自由エネルギー地形)を予測し、そのデータベースを作成する。当該領域との共同研究を積極的に行い、新たな研究を展開したい。